クロモエクセル [Chromoexcel]
クロモエクセルは、マウスの染色体上に、遺伝子が存在する場所と存在しない場所を、エクセルアートの手法を用いて描いた図です。併せて、私たちが研究している遺伝子トラップクローンの分布も示しています。 遺伝子トラップ [Gene Trap] というのは、マウスのES細胞 [Embryonic Stem Cell] を用いて、ES細胞で働いている(発現している)遺伝子を捕まえる方法です。
染色体 [Chromosome] は、私たちの体を構成している細胞の核の中に存在する糸状の物質です。お父さんとお母さんから受け継いだ遺伝情報が書き込まれています。ヒトの場合、1番から22番までの22対(44本)の常染色体と、男性はXY、女性はXXの性染色体、合計46本の染色体を持っています。マウスの場合は、常染色体が1番から19番までの19対(38本)なので、2本の性染色体を含めて合計40本の染色体を持っています。
遺伝情報は、染色体を構成しているDNAという物質に、A, G, C, Tという4文字で書き込まれています。マウスの場合、1番長い1番染色体(Chr.1)1本に約2億文字、全染色体(40本)の合計で約60億文字のA, G, C, Tが並んでいます。
私たちの体は、タンパク質 [Protein] という物質が基本となって構成されています。遺伝情報の中で、タンパク質の設計情報が書き込まれている部分を遺伝子 [Gene] と呼びます。DNAに書き込まれている遺伝情報は、DNAとよく似たRNAという物質に写し取られます。このイベントを転写 [transcription] と呼びます。RNAではA, G, C, Uという4文字が使われていて、DNAの「T」は、RNAに転写される時に「U」に変わります。RNAに写し取られたタンパク質の設計情報をもとにして、タンパク質が作られます。このイベントを翻訳 [translation] と呼びます。つまり、DNAに書き込まれている遺伝子の情報をもとにしてRNAが作られ、このRNAの情報をもとにしてタンパク質が合成されます。しかしながら、遺伝子の中には、タンパク質を作らないRNAの情報が書き込まれていることがあります。その場合、RNAそのものが生体内で重要な働き(機能)を持っていることが、近年の研究で分かってきました。
タンパク質の情報や、RNAの情報を持っている遺伝子の種類に関して、ヒトやマウスでは約5万個程度と考えられます。ただし、5万個の遺伝子が染色体上に均一に散らばっている訳ではなく、遺伝子がまとまって存在する領域もあれば、広い範囲にわたって遺伝子が全く存在しない領域もあります。その様な、遺伝子のばらつき具合を示したのがクロモエクセルです。
さて、ここからしばらく、遺伝子トラップの話をします。そのためには、遺伝子について、もう少し詳しい説明が必要になります。
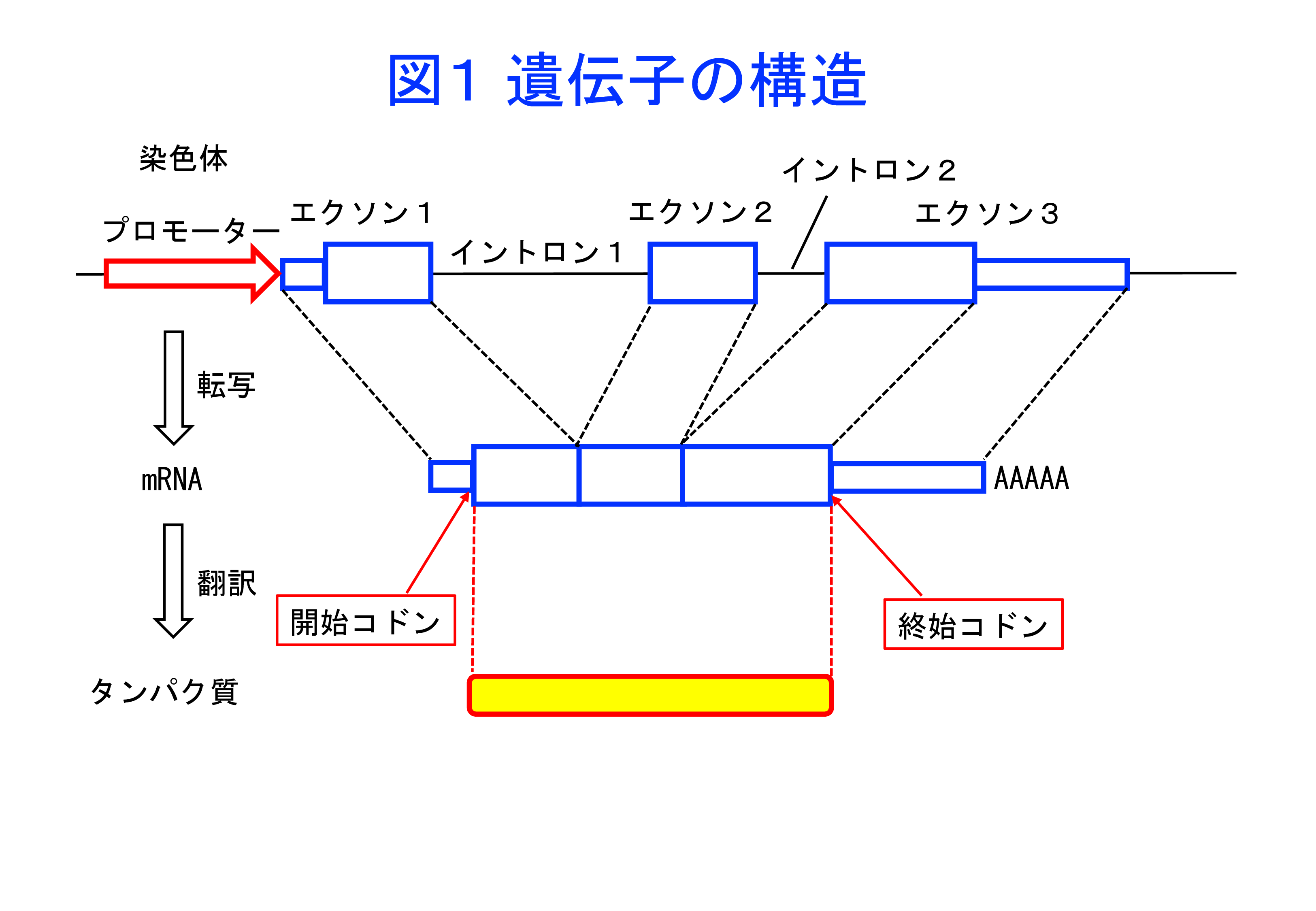
実は、遺伝子というのはA,G,C,Tで書かれた情報がひとかたまりで存在するとは限りません。RNAになる重要な部分だけではなく、間に余計なA, G. C. Tが並んでいることがあります。むしろ、その方が一般的です。 図1を見てください。タンパク質の情報が書き込まれているRNAをメッセンジャーRNA (mRNA)と呼びます。DNAとRNAにはA, G, C, T(U)の4文字で遺伝情報が書き込まれていますが、タンパク質は20種類のアミノ酸から構成されています。そして、RNAの文字3つがアミノ酸1つを意味します。このRNA3文字を「コドン」と呼びます。RNAに書かれている遺伝情報をタンパク質に翻訳する際に使われる暗号表がコドン表(図2)です。例えば、1文字目がA、2文字目がU、3文字目がGの場合、AUGというコドンはメチオニンというアミノ酸を意味します。ただし、このAUGは「開始コドン」とも呼ばれており、RNAの情報の中で、ここから翻訳を開始するという意味も持っています。また、翻訳の終わりを意味するコドンも存在します。それが終始コドンです。図1では、mRNAの開始コドンから終始コドンまでの情報がタンパク質に翻訳されることを示しています。
そして、染色体のDNA上には、mRNAの情報が書かれている部分と、そうではない部分があります。mRNAの情報が書かれている部分を「エクソン」と呼びます。遺伝子は一つのエクソンで構成されている場合もあるのですが、多くは二つ以上のエクソンから構成されています。二つのエクソンに挟まれた領域を「イントロン」と呼びます。図1では、エクソンが3つ、イントロンが2つの遺伝子を表示しています。 そして遺伝子には、それぞれの頭の部分に「プロモーター」と呼ばれる領域が存在します。プロモーターはmRNAを転写する際のエンジンの働きをします。プロモーターが無いと、mRNAへの転写は起こりません。そしてさらに、プロモーターにはどこでいつ働くかという情報が書き込まれています。例えば生まれた後に脳の細胞で働くという様な情報が書かれています。遺伝子は、このプロモーターの働き(機能)によって、どの細胞ではたらく(発現する)かが決まっています。 そのことを実際に調べることができます。その際使われるのがレポーター遺伝子です。レポーターというのは、報告するという様な意味になります。一番有名なレポーターは、オワンクラゲが持っている「光るタンパク質:GFP (Green Fluorescence Protein)」です。筋肉で働く遺伝子のプロモーター領域をとってきて、GFP遺伝子をつなげると、筋肉の細胞が光る様になります。脳で働く遺伝子のプロモーター領域にGFP遺伝子をつないだ場合は脳が光ります。 つまり、プロモーター領域は、パソコン付きのエンジンの様なものです。 さて、お待たせしました。ここから、遺伝子トラップの話をします。図3を見てください。 遺伝子トラップでは、トラップベクターという道具を使用します。トラップベクターにはレポーター遺伝子が組み込まれています。このレポーター遺伝子には、プロモーター、つまりエンジンが付いていません。このトラップベクターをマウスのES細胞の染色体に導入します。プロモーターが付いていませんので、染色体上の何も無いところに入っても、何も起きません。何かの遺伝子のプロモーターの下流に入った時に、そのプロモーターの働きでレポーター遺伝子のmRNAが作られ、そのmRNAの情報に従ってレポータータンパク質が作られます。トラップベクターに組み込まれているレポータータンパク質には、G418という抗生物質に抵抗するという性質があります。もともとのES細胞はG418という抗生物質が入っている培地の中では増えることができません。しかしながら、トラップベクターが何かの遺伝子の中に入って、レポーター遺伝子が働く(発現する)と、G418が入っている培地の中でも増えることができます。ES細胞が増えてできたかたまりをコロニーと呼びます。このコロニーを拾ってきて、バラバラにしてまた増やすことができます。そうやって作製した、同じ遺伝子構成を持つ細胞の集団をクローンと呼びます。遺伝子トラップの実験では、1回の実験で100個のコロニーが形成された場合、100種類のトラップクローンを作製することができます。 レポーター遺伝子を利用してES細胞で働いている遺伝子を捕まえた(トラップした)訳ですが、同時に、その遺伝子の働きを止めた(破壊した、ノックアウトした)ことになります。私たちは、この遺伝子トラップ法をさらに発展させて「可変型遺伝子トラップシステム」を構築しました(図4)。可変型というのは、一度トラップした遺伝子を、後で自由に改変することができる様な工夫をしたことを意味しています。その説明は省略します。 マウスのES細胞は、万能細胞と呼ばれることもあります。それは、このES細胞からマウス個体を造ることができるからです。ES細胞からキメラマウスを作製します。キメラというのは、異なる遺伝子構成を持つ2種類以上の細胞が混ざってできた個体を指します。このキメラマウスを野生型のマウスと交配することで、同じ遺伝子構成を持つマウスの集団(系統、マウスライン)を作製する事ができます。そしてトラップした遺伝子、つまりノックアウトした遺伝子が生体内でどんな働きをしているのかを、トラップマウスを使って調べる事ができます。 私たちは、可変型遺伝子トラップ法で作製したトラップクローンの情報を、EGTC(Database for the Exchangeable Gene Trap Clones)というデータベースで全世界に公開しています(図5)。 ES細胞として1,278クローンの情報を公開しています(図6)。その中の1,170クローンについて、トラップした遺伝子の染色体上の位置を決定しています。また、1,158クローン(90.6%)は既知遺伝子(名前が付いている遺伝子)をトラップしていたのですが、40クローン(3.1%)はトラップベクターが入った場所にはEST (Expressed Sequence Tag)と呼ばれるmRNAの一部と予想される遺伝子断片しか存在せず、80クローン(6.3%)は本当に何もない場所にトラップベクターが入っていました。一応New(新規遺伝子)と呼んでいます。この様な、EST及びNewに入る訳の分からないトラップクローンの解析を進めています。
また、514クローンについて、キメラマウスを作製し、マウスラインを樹立して、CARD R-BASEという、熊本大学 生命資源研究・支援センター 資源開発分野が構築したマウスバンクに寄託しています。 さて、世界各地で遺伝子トラップを大規模に行っている研究施設の情報を集めたIGTC (The International Gene Trap Consortium)という組織のデータベースがあり、EGTCもその中に入っています(図7)。IGTCには現在、ES細胞として121,703クローンの情報が登録されています。 さて、ある生物の細胞核の中にある遺伝子全ての情報を集めたものをゲノムと呼びます。ヒトの場合はヒトゲノム、マウスの場合はマウスゲノムです。膨大な数のA, G, T, Cが並んだゲノムの情報に、遺伝子などの情報を付加して見やすくしたデータベースをゲノムブラウザと呼んでいます。その中の一つであるUCSC Genome Browser on Mouse July 2007 (NCBI/mm9) Assemblyというデータベース(図8)には、IGTCの遺伝子トラップクローンの情報も書き込まれています。EGTCクローンのトラップベクターが入った場所を解析する際にも、このデータベースを利用しています。図8では、Bnc2(Basonuclin 2)という遺伝子をトラップした3個のEGTCクローン(Ayu21-W214, Ayu21-18, Ayu21-T516)が表示されています。この図は、マウスの4番染色体の真ん中あたり(419,073 bp)を拡大して見せています。また100 kbpを示す物差し(スケール)も表示されています。「bp」というのはbase pairの略なのですが、A, G, T, Cの数を表しています。「kbp」は1,000 bpの略です。つまり、419,073 bpは、約419 kbpです。 そして、EGTC及びIGTCのトラップクローンにおいてトラップベクターが入った場所がどの染色体に多いか、少ないかを示しているのが図9です。併せて、既知遺伝子(RefSeq)の分布を示しています。遺伝子(緑色)が一番多いのは7番染色体なのですが、トラップクローン(青色及び赤色)が多いのは2番、4番、11番染色体である事が分かります。 この、トラップクローンの分布と、遺伝子の分布をもっと細かく見たのがクロモエクセルです。エクセルアートという手法を利用しています。図10にその作り方を示しました。 エクセルというのは表計算ソフトですが、そのエクセルを使って描かれた絵のことをエクセルアートと呼びます。図8で示したマウスゲノムブラウザを使って、染色体の端から100 kbp毎に、遺伝子の数とトラップクローンの数を調べます。遺伝子が1個でもあれば水色、なければ無色です。また、各染色体のセントロメア側末端(0M)から3 Mbp (3,000,000 bp)はまだシーケンスが解読されていない領域でGapと呼ばれています。この部分は灰色にします。次に、トラップクローンが1個も無ければ無色、1〜19個あれば橙色、20個以上あれば赤色です。遺伝子もトラップクローンも、100 kbpで1列x3行使用します。図10はマウスの染色体の中で一番短い19番染色体(Chr.19)のクロモエクセルです。7Mあたりの拡大図も載せています。赤丸で示しているのは、遺伝子が1個も無いのに遺伝子トラップクローンが20個以上集積している領域(TCAA: Trap Clone Accumulated Area)です。
クロモエクセルを見ると、広い範囲に渡って遺伝子が全く無い領域(遺伝子砂漠)が結構あることがわかると思います。また逆に、遺伝子はあるのに遺伝子トラップクローンが全く無い領域も結構あることが分かると思います。遺伝子トラップというのは、主にES細胞で働いている(発現している)遺伝子を捕まえる方法ですので、遺伝子はあるのに遺伝子トラップクローンが全く無い領域は、その遺伝子がES細胞では働いていないことを示唆しています。逆に、遺伝子は無いのに遺伝子トラップクローンが数多く存在する領域というのは、私たちがまだ知らない遺伝子または遺伝子の様なものが存在することを示唆しています。